-
重叠基因 编辑
中文名:重叠基因
外文名:overlappinggene(s)
所属学科:生物学
别名:嵌套基因(nestedgenes)
定义:两个或两个以上的基因共有一段DNA序列,或是指一段DNA序列成为两个或两个以上基因的组成部分
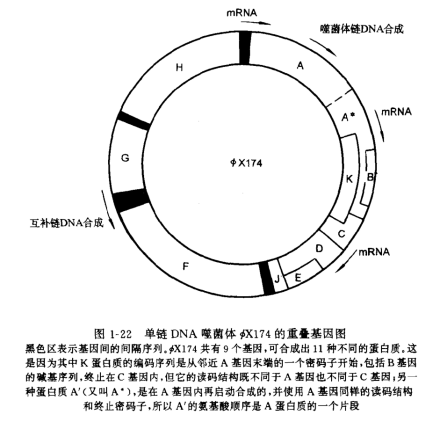
重叠基因 是在1977年发现的。早在1913年A.H.斯特蒂文特已在果蝇中证明了基因在染色体上作线状排列,50年代对基因精细结构和顺反位置效应等研究的结果也说明基因在染色体上是一个接着一个排列而并不重叠。但是1977年F.桑格在测定噬菌体ΦX174的DNA的全部核苷酸序列时,却意外地发现基因D中包含着基因E。基因E的第一个密码子(见遗传密码)从基因D的中央的一个密码子TAT的中间开始,因此两个部分重叠的基因所编码的两个蛋白质非但大小不等,而且 氨基酸 也不相同。在某些真核生物,如果蝇、人、秀丽新小杆线虫中都发现有重叠基因。
也是在1977年发现的,它是内部包含一段或几段最后不出现在成熟的mRNA中的片段的基因。这些不出现在成熟的mRNA中的片段称为内含子,出现在成熟的mRNA中的片段则称为外显子。例如下面这一基因,有三个外显子和两个内含子。在几种哺乳动物的核基因、酵母菌的线粒体基因以及某些感染真核生物的病毒中都发现了断裂的基因。内含子的功用以及转录后的加工机制是真核生物分子 遗传学 的一个吸引人的课题。
(见转座因子)首先于40年代中在玉米中由B.麦克林托克发现,当时并没有受到重视。60年代末在细菌中发现一类称为插入序列的可以转移位置的 遗传因子 IS,它们本身没有表型效应,可是在插入别的基因中间时能引起插入突变。70年代早期又发现细菌质粒上的某些抗药性基因可以转移位置。细菌中的这类转座子(Tn)到80年代已经发现不下20种,它们分别带有不同的抗药性基因,能在不同的复制子之间转移位置,例如从质粒转移到染色体、噬菌体以及别的质粒上等。当他们转移到某一基因中间时,便引起一个插入突变。类似于细菌转座子的可以转移位置的遗传因子在玉米以外的真核生物中也已经发现,例如酵母菌中的接合因子基因,以及果蝇白眼基因中的 转座因子 等。转座因子的研究也已成为分子遗传学中的一个重要方面。
功能、类别和数目到目前为止在果蝇中已经发现的基因不下于1000个,在 大肠杆菌 中已经定位的基因大约也有1000个,由基因决定的性状虽然千差万别,但是许多基因的原初功能却基本相同。
1945年G.W.比德尔通过对脉孢菌的研究,提出了一个基因一种酶假设,认为基因的原初功能都是决定蛋白质的一级结构(即编码组成肽链的氨基酸序列)。这一假设在50年代得到充分的验证。
重叠基因(overlapping gene):指两个或两个以上的结构基因共同一段DNA顺序的现象。
原核生物和一些病毒或噬菌体的基因组比较小,核苷酸对是极其有限的,那么怎样有效地利用这些有限碱基来编码更多的遗传信息呢?在生物中存在着一种十分巧妙的机制——重叠基因 (overlapping gene)。它的道理就像我们古代的回文诗一样,如: 莲人在绿杨津 采 一 玉漱声歌新阙 其读法是:采莲人在绿杨津,在绿杨津一阙新,一阙新歌声漱玉,歌声漱玉采莲人。本来需要28个字才能表达完的一首诗,现在利用前后两句之间的几个字的重叠,结果只用了14个字就完成了。重叠基因也正是如此,利用碱基的重叠来编码更多的信息。
重叠基因是在1977年首先发现的,当时美国著名的科学家SAnger建立了测序方法,他就用这种测序方法对环状单链的噬菌体F×174进行了测序。结果测出其基因组由5386个核苷酸组成,共有11个基因,构成3个转录单位,由3个启动子(pA,pB,pD)启动。(图10-4)基因的产物都已被分离,它们所含的氨基酸已远远地超过了5386个碱基所能编码的量,即F-174含有的5386Nt最多能编码1795个氨基酸,若每个氨基酸的平均分子量为110,则总的蛋白质分子量为197,000D,但实际测出的蛋白质总分子量却为262,000D。将全部DNA顺序和蛋白质的氨基酸顺序进行比较,发现B基因在A0基因之中,K基因跨在A-C两基因的连接处,和A0基因的尾部,C基因的首部相重叠,E基因在D基因内部。类似的情况在别的噬菌体如G4、微小病毒和SV40也有发现。重叠基因不仅可经济利用基因组,而且可能起表达调控的作用 重叠基因仅在噬菌体和病毒中存在,在真核生物中尚未发现重叠基因。这可能因为前者基因组比较小,但又必须要编码一些维持其生命和繁殖的基因,在选择的压力下,保留了这种重叠基因的形式。在本世纪70年代以前,人们一直认为遗传物质是双链DNA,在上面排列的基因是连续的。RobERT and Sharp彻底改变了这一观念。他们以腺病毒作为实验对象,因为它的排列序列同其他高等动物很接近,包括人。结果发现它们的基因在DNA上的排列由一些不相关的片段隔开,是不连续的。他们的发现改变了科学家以往对进化的认识,对于现代生物学的基础研究以及生物进化论具有重要的奠基作用,对于肿瘤以及其他遗传性疾病的医学导向研究,亦具有特别重要的意义。真核生物的基因组十分复杂,DNA的含量也比原核生物的大得多。噬菌体由于基因组很小,但又要编码一些必不可少的蛋白,碱基显然不够用,这样不仅几乎所有的碱基都参加编码,而且在进化中还出现了“重叠基因”,以有限的基因编码更多的遗传信息。真核基因组正好相反,DNA十分富余,这样不仅无需“重叠基因”,而且很多序列不编码,如重复序列、间隔序列 (spacer) 和间插序列(intervening sequence) 即内含子(intron)等。但不编码并不等于没有功能。有的我们可能还不了解,如重复序列。间隔区和间插序列这两个概念是不同的,间隔区是指基因间不编码的部分,有的转录称转录间隔区(TS),有的不转录称为非转录间隔区(NTS)。间插序列是指基因内部不编码的区域,也称内含子,在初始转录本中存在此序列,但在加工后将被切除掉,所以常不作为翻译的信息。间隔区常常含有转录的启动子和其它上游调节序列。有的内含子也可以编码,如成熟酶和内切酶等。
在遗传学上通常将能编码蛋白质的基因称为结构基因。真核生物的结构基因是断裂的基因。一个断裂基因能够含有若干段编码序列,这些可以编码的序列称为外显子。在两个外显子之间被一段不编码的间隔序列隔开,这些间隔序列称为内含子。每个断裂基因在第一个和最后一个外显子的外侧各有一段非编码区,有人称其为侧翼序列。在侧翼序列上有一系列调控序列(图3-3)。
调控序列主要有以下几种:
①在5′端转录起始点上游约20~30个核苷酸的地方,有TATA框(TATA box)。TATA框是一个短的核苷酸序列,其碱基顺序为TATAATAAT。TATA框是启动子中的一个顺序,它是RNA聚合酶的重要的接触点,它能够使酶准确地识别转录的起始点并开始转录。当TATA框中的碱基顺序有所改变时,mRNA的转录就会从不正常的位置开始。
②在5′端转录起始点上游约70~80个核苷酸的地方,有CAAT框(CAAT box)。CAAT框是启动子中另一个短的核苷酸序列,其碱基顺序为GGCTCAATCT。CAAT框是RNA聚合酶的另一个结合点,它的作用还不很肯定,但一般认为它控制着转录的起始频率,而不影响转录的起始点。当这段顺序被改变后,mRNA的形成量会明显减少。
③在5′端转录起始点上游约100个核苷酸以远的位置,有些顺序可以起到增强转录活性的作用,它能使转录活性增强上百倍,因此被称为增强子。当这些顺序不存在时,可大大降低转录水平。研究表明,增强子通常有组织特异性,这是因为不同细胞核有不同的特异因子与增强子结合,从而对不同组织、器官的基因表达有不同的调控作用。例如,人类胰岛素基因 5′末端上游约250个核苷酸处有一组织特异性增强子,在胰岛素β细胞中有一种特异性蛋白因子,可以作用于这个区域以增强胰岛素基因的转录。在其他组织细胞中没有这种蛋白因子,所以也就没有此作用。这就是为什么胰岛素基因只有在胰岛素β细胞中才能很好表达的重要原因。
④在3′端终止密码的下游有一个核苷酸顺序为AATAAA,这一顺序可能对mRNA的加尾(mRNA尾部添加多聚A)有重要作用。这个顺序的下游是一个反向重复顺序。这个顺序经转录后可形成一个发卡结构(图3-4)。发卡结构阻碍了RNA聚合酶的移动。发卡结构末尾的一串U与转录模板DNA中的一串A之间,因形成的氢键结合力较弱,使mRNA 与DNA杂交部分的结合不稳定,mRNA就会从模板上脱落下来,同时,RNA聚合酶也从DNA上解离下来,转录终止。AATAAA顺序和它下游的反向重复顺序合称为终止子,是转录终止的信号。