-
非编码RNA 编辑
中文名:非编码RNA
外文名:Non-codingRNA
属性:RNA
包括:rRNA,tRNA,snRNA,snoRNA
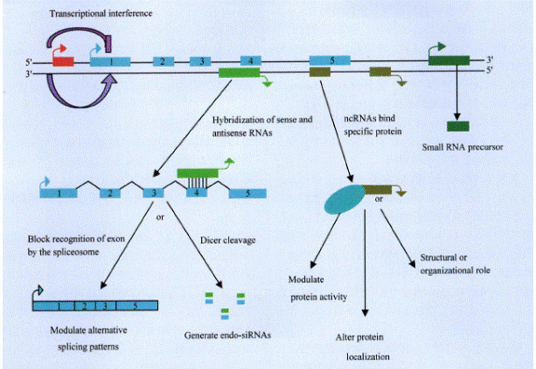
细胞中含量最高的是rRNA 和tRNA 这两种常见的非编码RNA。广义上的非编码RNA是包括这两种RNA的,研究的比较透彻。狭义上的非编码RNA往往不包括rRNA 和tRNA,因此在这里就不做介绍了。这里介绍的非编码RNA种类,是在狭义上的非编码RNA 的定义,即,不包括mRNA,tRNA 和rRNA 的其他RNA分子。在此主要介绍一下表达量相对比较高的snRNA、snoRNA 和作为研究热点的microRNA。
snRNA
snRNA 是small nuclear RNA的简称,也称作小核RNA。其功能是与蛋白因子结合形成小核核糖蛋白颗粒(small nuclear ribonucleo-protein particle,简称snRNPs),行使剪接mRNA的功能。snRNA 主要包括5 种:U1,U2,U4,U5 和U6。存在于所有snRNP 中的蛋白叫做通用蛋白,也称做sm 蛋白。通用蛋白和snRNA的结合位点已经研究清楚了。除U6 外,Sm蛋白可以结合到所有的其他snRNA 的保守序列AAU4-5GGA上,这段序列被称为Sm蛋白的结合位点,这也可以作为判断一个RNA是否是snRNA 的一个特征。
snoRNA
snoRNA 是最早在核仁发现的小RNA,称作小核仁RNA,最初发现它们的生物功能是用来修饰rRNA的。大多数小核仁RNA可以分为两类。一类是C Dbox snoRNA,这一类snoRNA是对RNA的碱基进行甲基化修饰的。其特点是含有4 个Box:Box C、Box D、BoxC’和BoxD’。事实上对C D box snoRNA 的预测已经有很多已经发表的软件,比如说snoscan。另一类是H/ACA box,这一类snoRNA 对RNA的碱基进行甲尿嘧啶化修饰。其特点是形成一个双stem,中间加一个loop 区,中间的loop 区中有一个boxH。而在尾部的tail 有个boxACA。由于boxH 和boxACA 的一级序列特征的界定比较松。比如BoxH 是ANANNA,很多A rich 的区域都能满足这个特征。而boxACA 是ACANNN,只有3 个碱基的明确信息。所以单单从一级序列特征上判断一个RNA 是否为snoRNA 是比较容易出错的。好在,HACA snoRNA 的二级结构是非常明确的。所以,用二级结构结合尾部的Box ACA的特征,联合判断一个RNA是否为HACA snoRNA 是切实可行的。
microRNA
MicroRNA 是一类21~23 nt 的小RNA,其前体大概是70~100 nt 左右,形成标准的stem 结构,加工后成为21~23 nt 的单链RNA。microRNA 的作用机制是与mRNA 互补,让mRNA 沉默或者降解。现在流行的RNAi 技术就是利用了细胞体内的这个机制,在体外人工加入类似microRNA 的smallRNA 来沉默对应的mRNA
circularRNA
环状RNA与已知的线状RNA不同,形成一个共价闭合的连续循环。在环状RNA中,RNA分子的3'和5'末端通常是连接在一起的。这种特性赋予了环状RNA许多特性,其中许多特性是最近才被识别出来的。
许多环状RNA来源于蛋白质编码基因,但是也有研究表明部分环状RNA可以编码蛋白质。因前期发现circRNA不具有编码蛋白的功能,因此它们被归类为非编码RNA。最近,一些环状RNA显示出了作为基因调节器的潜力。像许多其他非编码亚型一样,大多数环状RNA的生物学功能尚不清楚。
非编码RNA的工作主要分为两个方面,一是大规模的鉴定新的非编码RNA,二是通过各种方法研究非编码RNA的功能。大规模鉴定非编码RNA的方法可以分为用理论预测和实验发现两种。前者主要借助计算机,从已有的非编码RNA中提取特征信息,然后以特征信息做全基因组搜索,比较成功的预测软件有:预测snoRNA 的snoScan和snoGPS,预测tRNA 的tRNAScan,预测microRNA 的mirScan,另外如RNAz,RNAmotif 等也都是很好的非编码RNA预测工具。理论预测方法简便快捷,但是最终的确定还是依赖于实验,因此理论预测与实验验证通常是相辅相成的。
很多实验有鉴定非编码RNA的方法,这些方法被称作“RNomics”。RNomics 的核心在于构建非编码RNA 的cDNA 文库,根据长度不同,非编码RNA的cDNA文库可以主要分为小于50 nt,50~500 nt以及大于500 nt 3 种,另外对于大于500 nt 的还可以分为带polyA 尾巴和不带polyA 尾巴这两种。Ambion 公司、Invitrogen 公司以及Qiagen 公司都推出了可以用来构建非编码RNA 的cDNA 文库的试剂盒。得到非编码RNA的cDNA文库以后,有多种方法可以用来鉴定文库中的非编码RNA。比较传统的是克隆测序,这种方法工作量很大,且对低丰度的非编码RNA 检测效率较差,但是这种方法操作流程比较简单,且对仪器设备要求不高,成本相对较低,因此仍被广泛使用。随着测序技术的发展,得到非编码RNA的cDNA 文库以后,不用克隆就可以直接测序,这种方法简单快捷,工作量小,可以检测到大量低丰度的非编码RNA,但是对仪器要求较高,成本也很高,使用的测序仪主要有454 和solexa 两种。随着技术的改进和成本的降低,这种直接测序的方法将成为主流。
另外一种广泛使用的非编码RNA 检测技术,是2000 年以后发展起来的全基因组tiling 芯片技术,这种技术的核心在于构建高密度的覆盖全基因组的芯片,生产这种芯片的主要有affymetrix 公司和agilent 公司,这种技术的优点在于不用构建cDNA 文库,更不用做克隆,只要有分离纯化的非编码RNA就可以检测,操作简单,成本很低,可以检测到大量低丰度的非编码RNA,这种方法的缺点在于不能准确的确定非编码RNA的转录起始终止位点,也很难区分剪切加工产物。非编码RNA的功能研究主要集中在microRNA 和siRNA,主要原因在于这两种小RNA的功能作用方式,都是通过同目标基因进行碱基配对,寻找这些小RNA的靶点相对来讲比较容易;而对于长的非编码RNA来讲,它们往往要形成复杂的二级甚至是三级结构,预测其靶点比较困难。随着研究的深入,也有为数众多的长的非编码RNA的功能被鉴定出来,例如Xist 和Khps1 就属于长的带polyA 尾巴的非编码RNA。